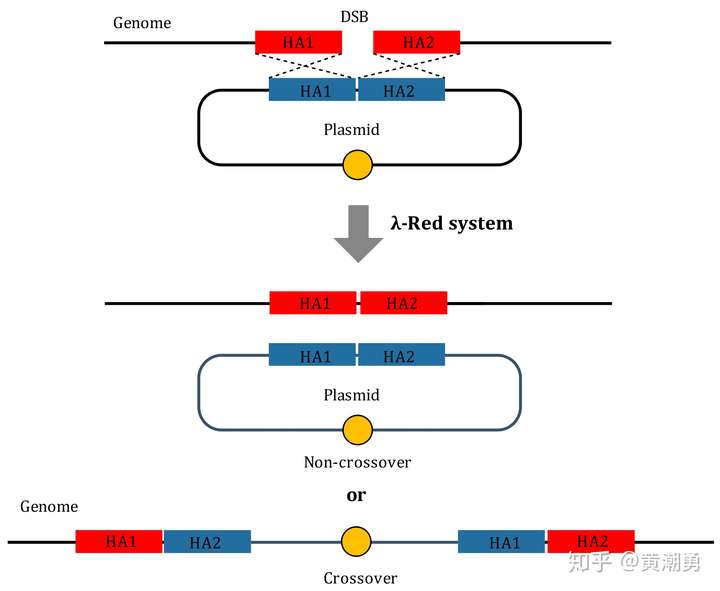
前面提到,将Donor DNA放在Plasmid#2上可以显著提高编辑效率,同时,这也让重组必须在基因组被切割之后发生。重组发生时,线性的基因组链侵入Plasmid#2,并以其为模板进行修复。这个修复的会产生两种结果,一种是我们想要的结果(如图5中的Non-crossover),另一种是整个Plasmid#2被整合到基因组上(如图5中的Crossover),这两种重组结果发生的概率基本相等,而用线性Donor DNA就不会出现这个问题。这是一个令人头疼的问题,难道鱼和熊掌真的不能兼得吗?

【图5】同源重组的两种结果 [2]
为了解决这个矛盾,我又想到了一个办法,就是把Donor DNA在基因编辑启动时从Plasmid#2上释放出来,以线性的形式参与重组。要达到这个效果,只需要在Donor DNA两侧加上基因组上的靶位点,如图6所示。这样一来,Cas9在切割基因组的同时,也会切割Plasmid#2并释放Donor DNA。由于基因组只有一个拷贝,而Plasmid#2有大约5个拷贝,因此只要控制好Cas9的诱导强度,就可以让基因组被切割,而Plasmid#2部分拷贝被切割,因此可以维持Plasmid#2的存在。
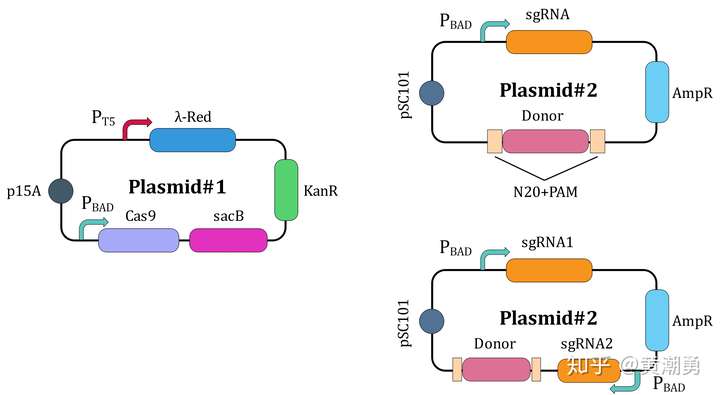
【图6】基因组编辑系统升级版四 [2]
按照这个策略,我测试的结果符合预期,Plasmid#2被整合到基因组上的情况不再发生了,平板上得到的菌落基本都是阳性的。大家可能发现了,图6中有两种Plasmid#2,一种只有一个sgRNA表达元件,一种有两个sgRNA表达元件。对于基因敲除、基因插入、基因替换、DNA大片段插入,只需要在基因组上切一刀就可以了,因此只需要表达一种sgRNA;而对于基因组大片段删除,则需要在基因组上切两刀,分别在要删除的大片段两端切割,这种情况就需要表达两种sgRNA。